Forschungsinteressen AG Kämper

Regulation der pathogenen und sexuellen Entwicklung in Ustilago maydis
Der Basisiomyzet Ustilago maydis ist einer der wichtigsten Modellorganismen zur Studie von Interaktionen zwischen Pflanze und Pathogenen. Ähnlich anderer Pflanzen- und Humanpathogenen ist U. maydis ein dimorpher Pilz, dessen haploide, hefeartige Zellen, genannt Sporidien, ausschließlich saprophytisch wachsen, während das filamentöse Dikaryon in seiner Entwicklung von der Wirtspflanze Mais abhängig ist. Nach Infektion der Pflanze induziert U. maydis die Bildung von Tumoren, in denen die Pilzhyphen differenzieren und diploide Sporen bilden. Das bedeutendste Merkmal des U. maydis-Systems ist, dass der Wechsel vom saprophytischen Wachstum zur biotrophen Lebensphase durch einen einzigen genetischen Schalter, den so genannten b-Lokus, kontrolliert wird, welcher für die zwei Transkriptionsfaktoren bE und bW kodiert.
Hauptinteresse unserer Arbeitsgruppe ist die Aufklärung der regulatorischen Prozesse, die zur Etablierung der pathogenen Wachstumsphase führen. Unser Hauptziel ist die Identifizierung und Charakterisierung von Genen, die durch den bE/bW Transkriptionsfaktor kontrolliert werden. Wir gehen davon aus das solche Gene eine entscheidende Rolle sowohl bei der morphologischen Umschaltung wie auch der pathogenen Entwicklung spielen.
Um eine flächendeckende Übersicht von b-abhängiger Genregulation und regulatorischen Netzwerken zu erhalten, haben wir DNA Microarrays etabliert, die eine parallele Beobachtung der Expression von etwa 6200 U. maydis Genen ermöglicht. Mit Hilfe dieser Gen Chip Technologie war es uns möglich, Änderungen der Genexpression nach Bildung eines aktiven bE/bW Heterodimers über einen Zeitraum von 12 Stunden zu beobachten und über 300 b-abhängige Gene zu identifizieren. Die bioinformatische Analyse dieser Gene ermöglichte die Visualisierung der verschiedenen Prozesse, die durch den b-Lokus kontrolliert werden. Eine große Anzahl der b-regulierten Gene haben Auswirkung auf den Zellzyklus, Mitose und DNA Replikation, was mit der Beobachtung übereinstimmt, dass die Zellteilung nach b-Induktion angehalten wird, bis der Pilz in die Pflanze eingedrungen ist. Andere Gene sind vermutlich in Synthese und Modifikation von Zellwandkomponenten verwickelt, was darauf hinweisen könnte, dass die Zellwand der hefeartigen Sporidien und des filamentösen Dikaryons sich in ihren Gesamtzusammensetzung unterscheiden.
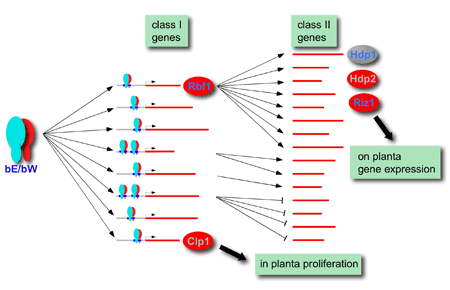
b-abhängiges regulatorisches Netzwerk. Klasse I Gene sind direkt durch bE/bW durch dessen Bindung an konservierte b-binde-Sequenzen (bbs) in den Promotorregionen reguliert. Die Mehrheit der indirket regulierten Klasse II Gene werden durch den b-abhängigen Transkriptionsfaktor Rbf1 kontrolliert. Clp1 wird unabhängig von Rbf1 exprimiert und wird für die Proliferation in der Pflanze benötigt. Die Expression der Transkriptionsfaktoren Hdp1, Hdp2 und Riz1 ist abhängig von Rbf1. Riz1 wird für die Induktion mehrerer Gene auf der Pflanzenoberfläche benötigt. In rot sind Proteine dargestellt, die obligatorisch für die pathogene Entwicklung sind, blaue Buchstaben geben an, das eine induzierte Expression zu einem Zellzyklusarrest führt.
Unter den b-regulierten Genen haben wir uns bisher auf Gene mit potentiell regulatorischen Funktionen fokussiert. Dies führte zur Identifikation von vier neuen Pathogenitätsgenen: riz1, rbf1 und hdp2, welche zwei Zinkfinger- und einen Homeodomänen-Transkriptionsfaktor kodieren, sowie clp1, welches für ein Protein mit unbekannter Funktion kodiert. Rbf1 wird sowohl für filamentöses Wachstum als auch für die pathogene Entwicklung benötigt. Microarry Experimente zeigten, dass dieser Transkriptionsfaktor eine Hauptrolle in der Expression eines Großteils der b-regulierten Gene spielt und damit eine zentrale Rolle im b-regulatorischen Netzwerk einnimmt. riz1, eines der Rbf1-regulierten Gene, wird für die Expression von Genen benötigt, die speziell auf der Pflanzenoberfläche induziert werden. Eine Großzahl der riz1-abhängigen Gene kodieren für U. maydis spezifische Proteine, die mutmaßlich sekretiert werden. Wir nehmen an, dass diese Proteine an der Hemmung von Pflanzenabwehrreaktionen sowie einer Umprogrammierung des Wirtes dienen. clp1 könnte für die Aufhebung des Zellzyklusarrests in der Pflanze erforderlich sein, da clp1 mutierte Stämme zwar in der Lage sind, die Pflanze zu infizieren, aber in ihrer weiteren Entwicklung noch vor der ersten Zellteilung blockiert sind. Wir werten zur Zeit Auwrikungen der verschiedenen Transkriptinosfaktoren aus und hoffen auf die Identifizierung von spezifischen Input- und Output Signalen und deren Verbindung zum pathogenen Entwicklungsprogramm.
Ein weiterer Fokus unserer Forschung ist das Verständnis eines Einflusses von Chromatin-vermittelter Transkriptionsregulation auf die pathogene Entwicklung. Wir haven bereits die Histondeazetylasen Hda1 und Rum1, ein Protein mit Ähnlichkeit zum humanen Retinoblastoma-bindenden Protein 2, identifizieren können, identifizieren können, die beide für die Bildung von Teliosporen benötigt werden. Daten aus Expressionsprofilen und Co-Immunopräzipitations-Experimenten platzieren Hda1 und Rum1 in einen gemeinsamen Komplex. Dieser Komplex wird für die Repression einer signifikanten Anzahl von genen benötigt, welche in Wildtypzellen im Tumor induziert sind. Interessanterweise sind einige dieser Gene nahe beieinander liegend, was ein weiterer Hinweis auf die Regulation auf Chromatinebene sein könnte.
Projekte
Folgende Projekte werden zur Zeit in der Arbeitsgruppe Kämper bearbeitet:
